無料でPythonの実行環境を使わせてもらえるGoogle Colaboratory。しかもGPUと12GBのメモリ、350GBのディスクまで使える環境であり、手元のPCよりハイスペックな人も多いだろう。
RユーザにとってはPythonだけでなくRでも使えたらいいのにと思うところである。そこで、この記事ではこの環境でRを使う方法を解説する。
目次
Rを使う4つの方法
実はGoogle ColaboratoryのインスタンスにはRの環境(RのバイナリとJupyter Kernel)がインストールされており、わずかな手順で使えるようになるのである。
参考までにインストールされているKernelの一覧はマジックコマンドから確認できる。
!jupyter kernelspec list
Available kernels:
ir /usr/local/share/jupyter/kernels/ir
python2 /usr/local/share/jupyter/kernels/python2
python3 /usr/local/share/jupyter/kernels/python3
swift /usr/local/share/jupyter/kernels/swift
Pythonはもちろん、R、Swiftもインストールされている。
スクリプトの実行
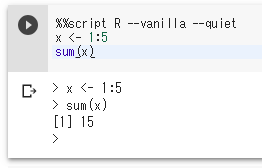
マジックコマンドの%%scriptを使う方法である。
Rに限らず、その言語がインスタンスにインストールされていれば使えるので知っておくといい(Perlなども使える)
%%script R --vanilla --quiet
x <- 1:5
sum(x)
--vanillaでなく--saveにすると最後の時点での.RDataを保存する。次回開くときに--restoreとするとその.RDataを読み込む。
%%script R --save --quiet
%%script R --save --restore --quiet
スクリプトの実行では、処理の途中まで実行して変数を確認したりプロットを画面で表示したりすることはできない。
一つのスクリプトで最後のアウトプットまで完了する必要があり、アドホックな用途には向かない。
単なるシェルの実行環境であって、Notebookではないのである。
Rのランタイムを使う(1)
Notebookとして使う(その場でプロットしたりオブジェクトを保存・再利用できる)ためにはランタイムを使うことになる。
この場合Notebookの中だけではできず、いったんipynb自体を書き換える必要がある。
- ipynbファイルをダウンロード
- 書き換え
- アップロード
既存のNotebookのipynbファイルをダウンロードする
書き換えるのは以下の部分
"kernelspec": {
"name": "python3",
"display_name": "Python 3"
},
以下のように書き換える。
"kernelspec": {
"name": "ir",
"display_name": "R"
},
新規Notebook作成から「アップロード」でこのファイルをアップロードする。
するとそのままNotebookとしてRのコマンドを使えるようになる。
この方法はKernelがインストールされているSwiftでも可能。ipynbのkernelspecの設定で
"kernelspec": {
"name": "swift",
"display_name": "Swift"
},
とすればOK。
Rのランタイム環境下ではマジックコマンドを使えない。シェルの実行やファイル転送ができなくなるので不便なところがある。
Rのランタイムを使う(2)
Notebookを新規作成する際のURLでlanguageパラメータを指定することでAvailable kernelsの中から任意のカーネルを採用できる。Rの場合は以下のURLになる。
https://colab.research.google.com/notebook#create=true&language=r
なおR以外にも以下の言語を指定できる(languageを指定しない場合はPython3)
- Swift: https://colab.research.google.com/notebook#create=true&language=swift
- Python2: https://colab.research.google.com/notebook#create=true&language=python2
Python環境と共存したまま使う
そこでマジックコマンドを使える状況下で、Python環境とも共存しながらRを使う方法を紹介する。
といっても以下のコマンド一発でOK。
%load_ext rpy2.ipython
rpy2モジュールを有効化すればいい。これでPythonのコードブロックもそのまま使うことができるし、マジックコマンドも使える。
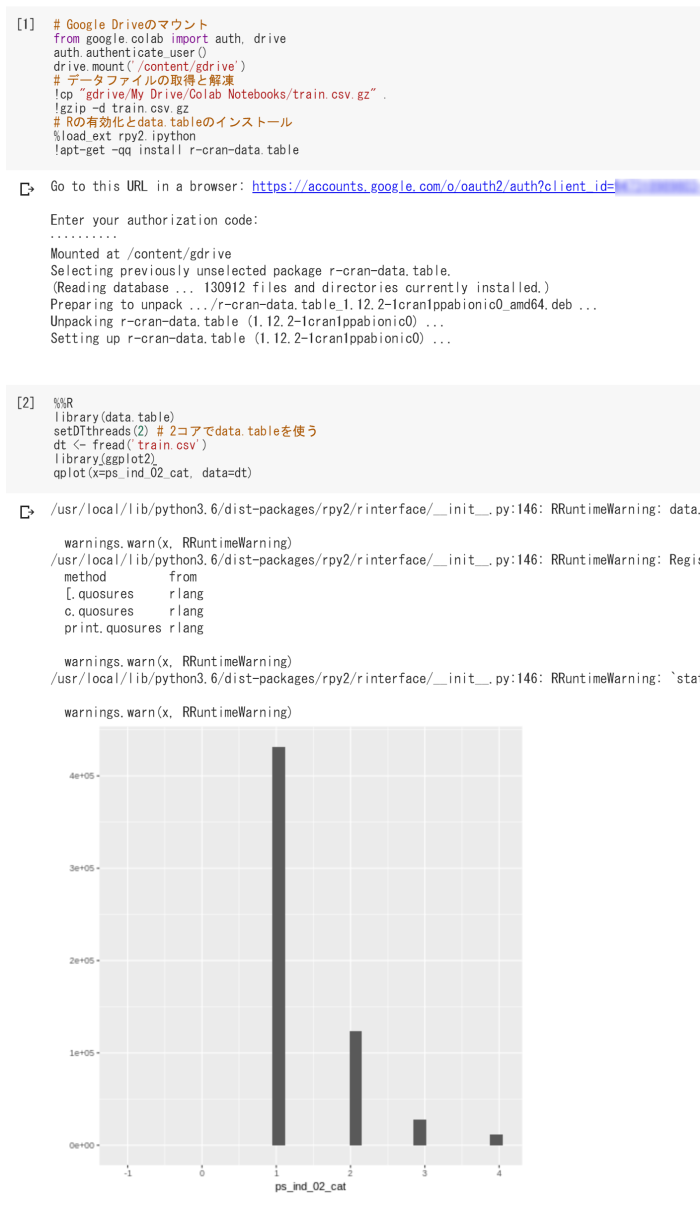
# Google Driveのマウント
from google.colab import auth, drive
auth.authenticate_user()
drive.mount('/content/gdrive')
# データファイルの取得と解凍
!cp "gdrive/My Drive/Colab Notebooks/train.csv.gz" .
#!gsutil cp gs://my_bucket/train.csv.gz . # Google Cloud Storageを使う場合
!gzip -d train.csv.gz
# Rの有効化とdata.tableのインストール
%load_ext rpy2.ipython
!apt-get -qq install r-cran-data.table
Rを実行したい場合、マジックコマンド%%Rで始まるR専用のコードブロックを作る。
%%R
library(data.table)
setDTthreads(2) # 2コアでdata.tableを使う
dt <- fread('train.csv')
library(ggplot2)
qplot(x=ps_ind_02_cat, data=dt)
このようになる。
- PythonでGoogle Driveをマウントをし
- マジックコマンドでファイルをコピー
- Rで読み込む
- 実行結果をGoogle Driveにコピー
一連の流れが可能になる。これは便利。
Pythonと変数をやりとりする
PythonからRに変数を渡す場合
%%R -i Python側の変数名
Rのコード
:
Python側のPandas DataFrameなどの変数名を指定すればその変数名のままRで使うことができる。ただしobject型は扱えない。つまり文字列やNoneを含むカラムがあるとエラーになる。
%%bigquery --project my-project df
SELECT ...
などマジックコマンドで取得した変数も使える。
RからPythonに変数を渡す場合
%%R -o R側の変数名
Rのコード
:
となる。
高速化
data.tableでマルチスレッド化
高速にデータフレームを扱えるdata.table、しかもマルチスレッドに対応しているので、これはマスト。
data.tableパッケージはインストールされていないので先にインストールする必要がある。
!apt-get -qq install r-cran-data.table
Google ColaboratoryではCPU2コア使えるので、しっかり明示する。
%%R
require(data.table)
setDTthreads(2)
GPUを使う
Google ColaboratoryといえばGPU。これをRで使えないものか・・・
少し手間はかかるが、使うことはできる。
BLAS
処理の内容(機能)ごとにさまざまなGPU対応ライブラリがあるが、もっとも簡単なのはBLASをGPU対応のものに入れ替えること。BLASを使う処理(大部分の行列処理)は多くあり、それらは軒並み高速化される。またBLASの入れ替えの場合はライブラリ固有の関数を覚える必要がない。非GPUと同じ関数で高速化できる。
GPU対応BLASをRで使うためには、
- GPU用のBLASの設定(nvblas.conf)をし、
- Rの実行時にGPU用のBLASを読み込むようにLD_PRELOADを指定する
- 同時に設定ファイルを指定する変数NVBLAS_CONFIG_FILEも指定する
これらをまとめた以下のコマンドを実行するとRコマンドそのものをGPU/BLAS対応のものに置き換えることができる
%%bash
echo "NVBLAS_LOGFILE nvblas.log
NVBLAS_CPU_BLAS_LIB /usr/lib/x86_64-linux-gnu/libopenblas.so
NVBLAS_GPU_LIST ALL" > /etc/nvblas.conf
mv /usr/local/bin/R /usr/local/bin/R_
echo '#!/bin/sh
LD_PRELOAD=/usr/local/cuda-10.0/targets/x86_64-linux/lib/libnvblas.so NVBLAS_CONFIG_FILE=/etc/nvblas.conf /usr/local/bin/R_ "$@"' > /usr/local/bin/R
chmod +x /usr/local/bin/R
現時点でGoogle ColaboratoryのRでGPU対応BLASを使うためには上記のスクリプトとしての実行しかできない。
というのもrpy2ではR実行時の環境変数を指定することができない。またランタイムを使おうにもNotebook起動時に環境変数として設定されている必要がある。Notebook起動後しかアクセスできないGoogle Colaboratoryでは不可能(自前のNotebookであれば可能だが)。LD_PRELOADを指定する手段が上記のスクリプトしかないのである。
BLAS以外のGPU利用例
BLASを使う計算以外は個別のライブラリごとにBLAS対応させることになる。
ここではXGBoostをGPU対応させる方法を紹介する。
GPU対応のXGBoostをコンパイル、インストールする。
%%bash --bg
git clone --recursive https://github.com/dmlc/xgboost
cd xgboost
mkdir build
cd build
# マルチGPUではないので -DUSE_NCCL=ONは指定しない
cmake .. -DUSE_CUDA=ON -DR_LIB=ON
# コア数2でコンパイル
make -j2
make install -j
xgb.train()実行時にパラメータでtree_method = 'gpu_hist'を指定すればGPUを使えるのだが・・・
Will run very slowly on GPUs older than Pascal architecture.
https://xgboost.readthedocs.io/en/latest/gpu/
Google ColaboratoryのGPUはK80(Kepler)であり、つまり遅くなるということ。Google ColaboratoryのGPUインスタンスはXGBoostでは不適ということになる。
GPUの利用については課題が残るものの、使えないことはない。今後のrpyライブラリやGPUの更新に期待したい。
[公開日:2019年6月10日]データの加工や分析で使うRの使い方 の記事一覧